文章图片
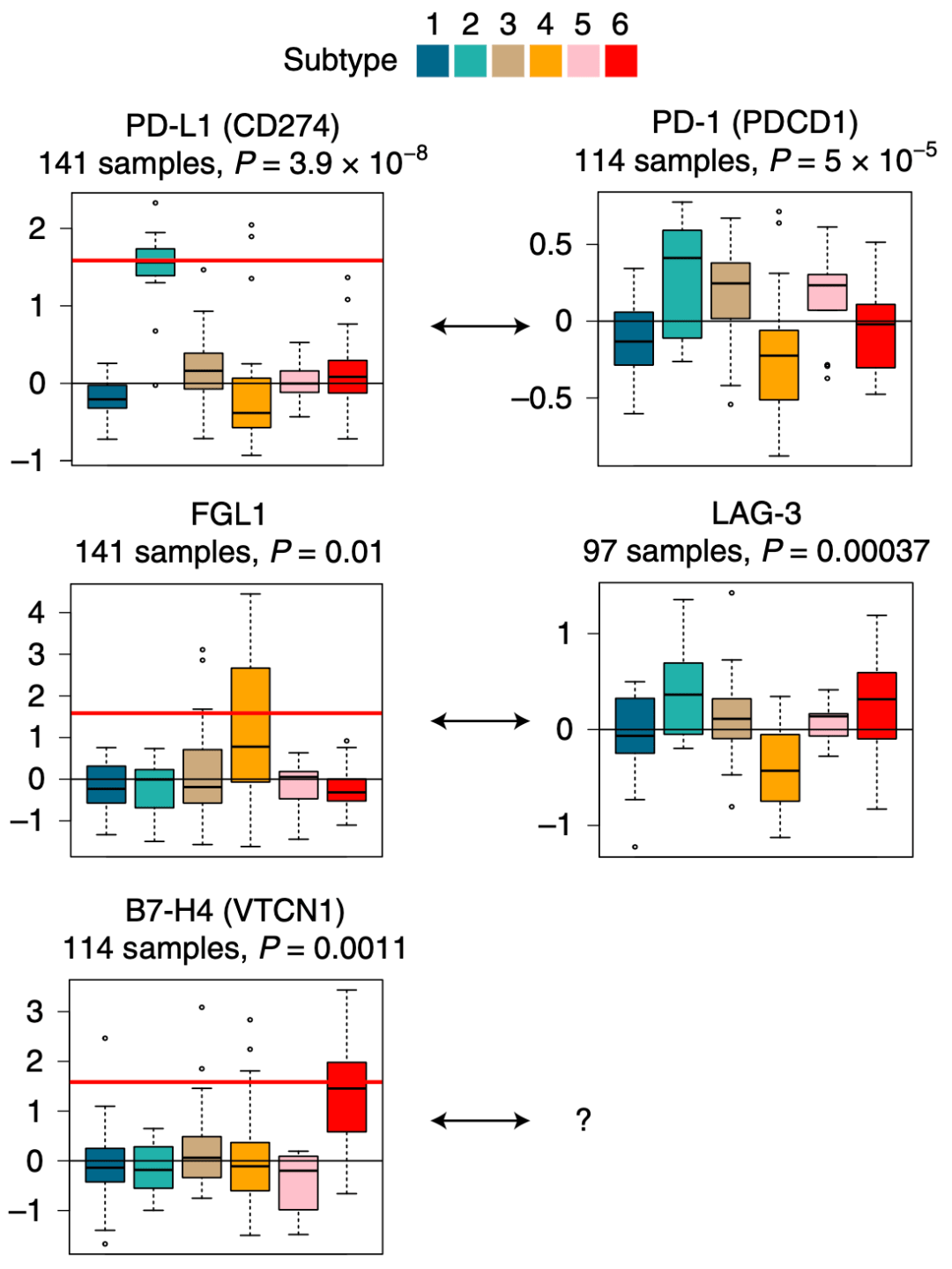
▲ 抑制性受体(IRs)及其配体在6个NSCLC亚型中的蛋白表达水平 。 左列箱型图中的红线标明了可视为表达水平异常的阈值
近期研究发现 , FGL1由肿瘤细胞分泌 , 它是LAG-3的高亲和性配体 , 可带来FGL1-LAG-3介导的T细胞抑制[6] 。 B7-H4是一种防止自体免疫的检查点蛋白[7] , 在小鼠模型中 , 靶向B7-H4可以减少肿瘤生长速度和肺部转移灶形成[8] 。 这些发现有力地支持了本研究对蛋白组亚型的免疫检查点分析 , 说明此类分析对于免疫检查点抑制剂的选择有重要预测价值 。
Lehti?团队对亚型4中的FGL1产生了兴趣 。 它背后是什么机理呢?
Lehti?团队通过对蛋白及转录组的相关性分析发现 ,肿瘤抑制因子STK11/LKB1和FGL1这两者的蛋白表达水平具有很强的负相关 , 但相应的mRNA水平却没有这种相关性 , 说明STK11还受到转录后调控 。
蛋白和mRNA表达均与FGL1相关性最高的是CPS1 。 CPS1是一种线粒体尿素循环酶 , 已有研究证明它在癌症中表达上调 , 是通过STK11失活后的AMPK-mTOR信号通路[9] 。
通常情况下FGL1和CPS1仅在肝脏中表达[6] , 但本研究数据表明 ,在NSCLC中STK11失活导致了这两个基因的转录增加 , 导致细胞增殖加快、mTOR信号通路失控以及免疫逃逸 。 这个发现提示了一种新治疗策略: 针对属于亚型4的病例 , 或许可以使用LAG-3/FGL1免疫检查点抑制剂和mTOR抑制剂联合治疗 。
在NSCLC的攻坚战中 , 除早期筛查之外 , 预测治疗效果和优化治疗组合是最关键的临床需求 , 而这离不开对该疾病更加系统的理解 。
Lehti?团队这个研究运用的蛋白组学分析 , 鉴别出了NSCLC的不同亚型 , 并分别确认了相应的特异性蛋白分子、信号通路和免疫表型 , 有望为更精确有效的肺癌治疗提供重要帮助 。

文章图片
参考文献:
[2] Cancer Genome Atlas Research Network. Comprehensive molecular profiling of lung adenocarcinoma [published correction appears in Nature. 2014 Oct 9;514(7521):262. Rogers, K [corrected to Rodgers, K]] [published correction appears in Nature. 2018 Jul;559(7715):E12]. Nature. 2014;511(7511):543-550. doi:10.1038/nature13385
[3] Charoentong P, Finotello F, Angelova M, et al. Pan-cancer Immunogenomic Analyses Reveal Genotype-Immunophenotype Relationships and Predictors of Response to Checkpoint Blockade. Cell Rep. 2017;18(1):248-262. doi:10.1016/j.celrep.2016.12.019
[6] Wang J, Sanmamed MF, Datar I, et al. Fibrinogen-like Protein 1 Is a Major Immune Inhibitory Ligand of LAG-3. Cell. 2019;176(1-2):334-347.e12. doi:10.1016/j.cell.2018.11.010
[8] Jeon H, Vigdorovich V, Garrett-Thomson SC, et al. Structure and cancer immunotherapy of the B7 family member B7x. Cell Rep. 2014;9(3):1089-1098. doi:10.1016/j.celrep.2014.09.053
[9] K Kim J, Hu Z, Cai L, et al. CPS1 maintains pyrimidine pools and DNA synthesis in KRAS/LKB1-mutant lung cancer cells [published correction appears in Nature. 2019 May;569(7756):E4]. Nature. 2017;546(7656):168-172. doi:10.1038/nature22359
- 卫生室|《乡村医生诊疗口袋书(先行本)》面世
- 解读|《浙江省医院门诊管理办法》政策解读
- 廉东波|《健谈》观点丨浅表性胃炎三五年复查一次 萎缩性胃炎一年一查
- 家长|杭州《青少年皮肤健康状况调研》发布,近半青少年受皮肤疾病困扰
- 公报,监测,国民,体质|一图读懂《第五次国民体质监测公报》
- 作者:鲍秀兰专家团队本文转载自“儿科医生鲍秀兰”(xiehebaoxiulan)来源:金...|孩子老喊腿疼,是长高的预兆吗?如果有这些症状别大意
- 感冒|《气血运行影响心脏功能——心悸篇》
- 幽门螺杆菌感染|《健谈》观点丨突然消瘦、胃痛、黑便,应及时做胃镜检查
- 数据|《胃肠病学》:减肥防早发肠癌,要从20岁开始!科学家发现,20、30岁和约45岁时肥胖,与结直肠癌发病风险上升88-156%有关
- 医学|科普连载:《氢气医学人群试验》(漫画版)孙学军教授 序言
