在肿瘤治疗中 , 使用单药治疗容易导致患者耐药 , 而肿瘤药物的联合治疗对克服耐药性 , 加强药物疗效 , 降低单药剂量毒性 , 以及扩大药物适应症有非常大的帮助 。 但是我们对不同药物联用的方法和疗效的了解远远不够 , 潜在的药物组合数量大大超出了临床试验的数量 。
目前关于联合用药的各种研究也存在着各种各样的不足 , 比如说 , 检测的靶向药物以及组合方式很少 , 或者是检测时药物浓度范围的选取不太理想等等 。 此外 , 以前的研究使用的细胞系数量也不够多 , 就很难将药物组合的活性和分子背景系统性地联系起来 。
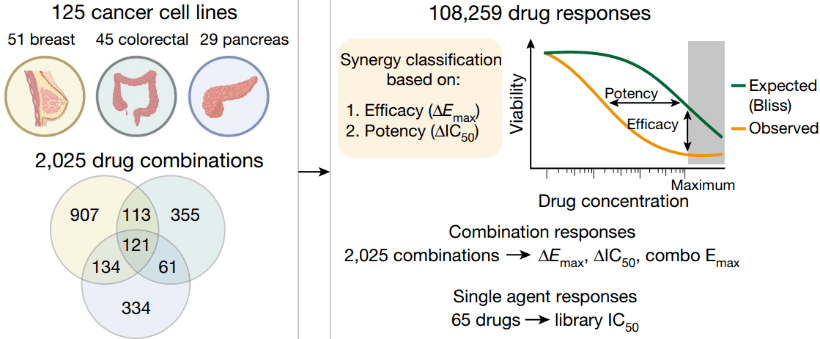
近日 , 由英国 Wellcome Sanger研究所Mathew Garnett领导的研究团队在著名期刊《自然》上发表重要研究成果[1] 。研究团队使用癌症药物敏感性基因组学(GDSC)细胞系筛选平台来系统性评估2025种双药组合在125个细胞系中的治疗效果 , 他们创建了目前世界上最大的开放数据库之一 , 并对各种有效药物联合进行了系统性的多组学分析 。 这项研究的目的不在于某个特异的或者创新性的靶标 , 而是为了将这个工作量巨大的数据库 , 还有对这个数据库的多组学研究方法分享给有需要的人 。

文章图片
论文首页截图
文章图片
图1:2025种双药组合在乳腺癌、结直肠癌和胰腺癌中的筛选
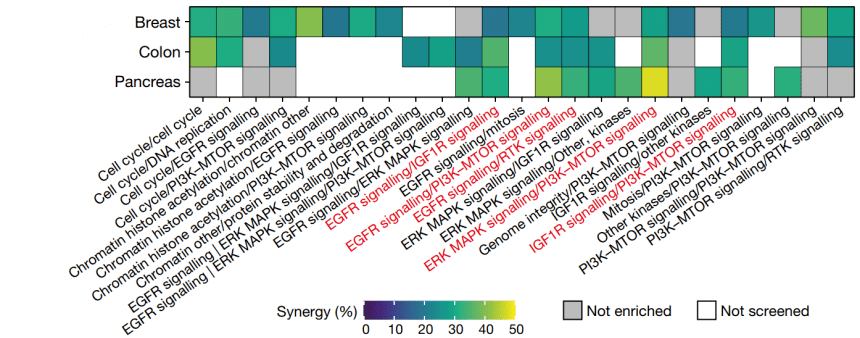
同时在通路水平上 ,18%的通路至少在一个癌种中具有显著协同作用 , 而且有五个通路组合在胰腺癌、乳腺癌和结肠癌三个癌种中都具有协同作用 , 也就是下图中标红的五个通路 。
文章图片
图2: 25个通路在至少一个癌种中具有显著协同作用
研究人员还结合药物基因组学分析(GDSCTools)来确定这些药物组合中的生物标志物 , 用于分析分子特征和药物反应之间的关联 。 这一分析结果可以直观看出不同细胞对药物治疗的反应是非常不同的 , 这些数据也可以为临床治疗的开发提供帮助 。
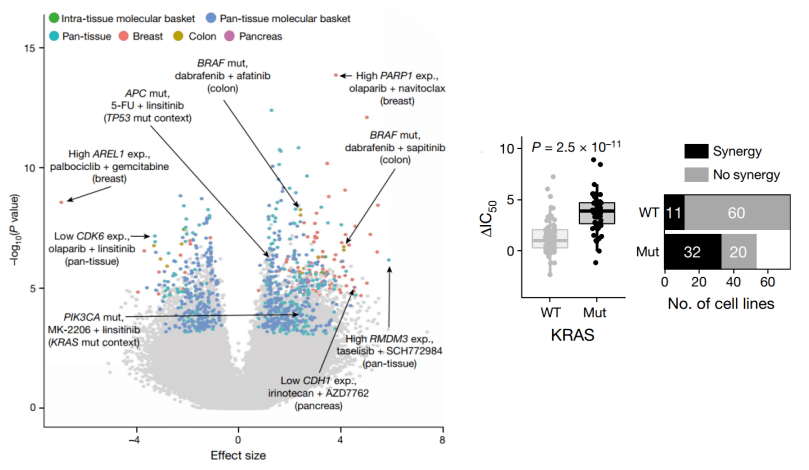
紧接着 , 研究人员将单药和药物组合的生物标志物覆盖在IntAct蛋白质-蛋白质相互作用网络上 , 发现许多药物协同组合可以与生物标志物相关联 。 例如 大名鼎鼎的KRAS突变就与曲美替尼(MEK1/2抑制剂)和MK-2206(AKT1/2抑制剂)联用的敏感性显著相关 。 这一分析结果不仅扩展了已知的临床生物标志物的研究 , 甚至还能应用到临床前和临床随访中的患者分层当中去 。
文章图片
图3:左图 , 与ΔIC50相关的生物标志物火山图;右图 , KRAS突变与曲美替尼+MK-2206组合反应相关
同时 , 研究人员比较了三个癌症亚型(基底样乳腺癌、MSS结肠癌或KRAS突变型结肠癌)中的药物组合 , 与来自同一癌种其他亚型的组合协同率 , 通过筛选得到了75种有潜力的药物组合 。
研究人员又进一步比较了这些药物组合与正在进行的临床试验 , 确定了11种组合 。 比如 ,他们发现了顺铂与吉西他滨(一种嘧啶抗代谢物)或 MK-1775(WEE1和PLK1抑制剂)的药物组合在基底样乳腺癌中联用效果非凡 , 而这两种药物组合都应用到了三阴性乳腺癌的临床试验中 。 也就是说 , 这个数据库能够为临床试验提供数据支持 , 给不符合标准用药指南的病人带来新的药物联用的希望 。
- 空调|《自然》子刊:空调续命,有了科学依据!中科院团队用两种小型恒温动物揭示,体温是比代谢率更重要的寿命决定因素丨科学大发现
- 积分|“核麟麟”带你闯关、答题、拿积分!《核平精英》上线啦!
- 乳腺癌|充满前景的新型药物可防止乳腺癌细胞苏醒
- 总体|《2021年成都市国民体质监测公报》公布 达标率实现6连升
- 衍生|新品 | 衍生蓓儿撼宁呵护健康,无惧换季感冒!
- 治疗|儿童肿瘤药物何以被人遗忘
- 技术|国家卫健委发文|北医三院骨科关节外科牵头制定的《髋膝关节置换术操作规范(2022年版)》正式实施
- 本文转自:西部网胰岛素是部分糖尿病患者的常用降糖药物|胰岛素注射技术全攻略 小细节避免大危害
- 疫情|注意!核酸检测指南有重大调整
-  |教学课丨《过去已成为过去》网红舞曲分解教学
